安迪·埃文斯发现,分子计算系统可以改善疾病诊断,甚至可以侵入活细胞
“计算机病毒”一词是由20世纪90年代初的美国计算机科学家提出的Len期刊将他的注意力转移到与之同名的最阴险的生物之一:艾滋病毒。这位南加州大学(University of Southern California)的研究人员早在上世纪70年代就已经与人合作发明了一种算法,至今仍为安全的在线交易提供支持,他甚至还为1992年的黑客电影担任过顾问运动鞋.但尽管他名声在外,没有人愿意听他对艾滋病的看法。
为了“更好地学习真正的艾滋病毒研究人员的语言”,阿德曼花了一个夏天在他大学的分子生物学实验室里。结果是一个新的科学领域,完全分散了他对艾滋病毒的注意力。“我认为DNA是一个数学对象——它只是一串字符,”阿德曼回忆道。“我把聚合酶链式反应等技术视为计算。”
当他早期的实验表明DNA可以计算,甚至可能被用来解决棘手的数学问题——包括破解密码方案——比硅更好时,阿德曼建立了自己的实验室,专注于DNA计算。尽管他的探索为他赢得了“DNA计算之父”的称号,但十年后,他得出结论,DNA不会取代硅来解决复杂的数学问题。阿德曼在2002年发表了关于这一主题的最后一篇论文收回了来自青少年领域的活跃研究。
被父亲抛弃,局限性暴露,DNA计算似乎陷入了危机。但多亏了阿德曼实验室奠定的部分基础,DNA计算和分子编程现在意味着人类可以理性地设计生化系统的控制,甚至是生物体内部的生化系统。
计算,但不是我们所知道的
DNA计算在1994年出现了爆炸性的发展,当时Adleman证明了DNA链可以解决“旅行推销员的问题。1挑战是在各个城市之间找到一条路线,每个城市只经过一次。因为每增加一个城市,可能答案的数量就会呈指数增长,这个问题很快就会耗尽传统计算机的资源。
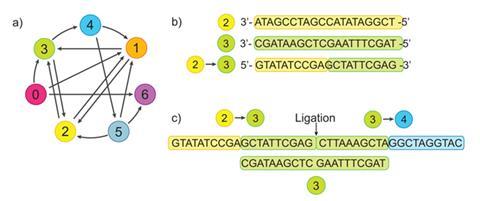
阿德曼用19个不同序列的DNA链来表示地图,每个序列有20个核苷酸碱基长。其中7个序列表示城市,12个序列表示城市之间可能的路径。并不是每个城市都与其他城市相连。每条路径链的前10个核苷酸与代表该路径连接的第一个城市的序列的后10个核苷酸互补。一条路径链的最后10个核苷酸与代表该路径连接的第二个城市的序列的前10个核苷酸互补。因此,每条路径都可以与两个城市配对成DNA双链,而除了第一个和最后一个城市之外的每个城市同样也会与两条路径配对。由于DNA连接酶的存在,每个阶段都可以连接在一起,成为解决问题的“旅程”序列。
在七天的实验室工作中,阿德曼将不同的城市和道路混合在一起,得到了一组精选的组合旅行。他用酶驱动的聚合酶链反应(PCR)扩增出更多连接目标起始城市和终点城市的路径副本,并纯化出没有访问过每个城市一次的路径副本。再次用PCR扩增剩下的几条DNA,得到足够的DNA,然后可以在凝胶色谱仪上读出条带。
在这50皮摩尔的反应中大约是3 × 1013每个起始寡核苷酸的副本都可用于并行计算。如果将每个绑定都视为一个计算操作,在为期一周的实验中约为4 × 1014每一次操作都只使用少量的能量。在那个时候,最好的硅计算机可以做10个左右12每秒操作数,或约7 × 1014两小时后。随着进一步的改进似乎有可能实现,媒体和科学家都兴奋不已。
然而,扩大规模被证明是棘手的。2002年,阿德曼和他的团队发表了他们最大的DNA计算,涉及超过100万个不同的序列。2当时,他们写道,这可能是迄今为止通过非电子手段解决的最大问题。这种规模的问题似乎超出了人类独立计算的正常范围。”
然而,美国德克萨斯大学奥斯汀分校生物化学家安迪·艾灵顿后来强调,如果那些没有帮助的人得到电子计算机的帮助,这个问题就可以“小菜一碟”。3.作为并行DNA计算的严厉批评者,艾灵顿曾称其为“技术失败的一个很好的例子”。它的衰落源于不必要的互动。每当出现错误配对时,系统就会将信息转换为无意义的信息。当你在百万个分子中寻找一个“正确答案”时,这可能是致命的。艾灵顿说:“很明显,要想让DNA的准确性达到普通手持计算器的水平,就需要在分子工程方面取得真正非凡的成就。”
尝试不平凡
到那时Erik Winfree他在美国帕萨迪纳市的加州理工学院(Caltech)攻读博士学位时,正在与阿德曼合作,攻读DNA瓦等结构的自组装编程,目前他在那里有自己的团队。那次经历向他展示了DNA如何可以执行一种最初比较温和,甚至可能不太熟悉的计算类型。在此基础上,他的加州理工学院团队将开发DNA链交换伙伴的化学反应网络,利用一种被称为“足尖点介导的链位移”的现有概念来制造电路。
这个立足点是一个核苷酸序列,悬在两个结合的长链上,长度不均匀。如果一个进入的寡核苷酸有正确的序列与较长的伴侣结合,包括额外的悬垂的“立足点”,它可以迅速取代较短的伴侣。使用这种无酶,非共价的方法,Winfree的团队在2006年发表了详细的与,或,而不是电路——最基本的逻辑要素。4
当两个输入信号都接通时,与门产生输出信号。加州理工学院团队的版本由三部分组成,两条较短的线连接到一条较长的线上。第一寡核苷酸输入通过脚尖位移去除其中一条短链。这为剩下的一对链打开了一个立足点,其中一条是荧光的,另一条是荧光猝灭的。第二个输入的寡核苷酸将它们分离,最终产生荧光,即电路的读数。
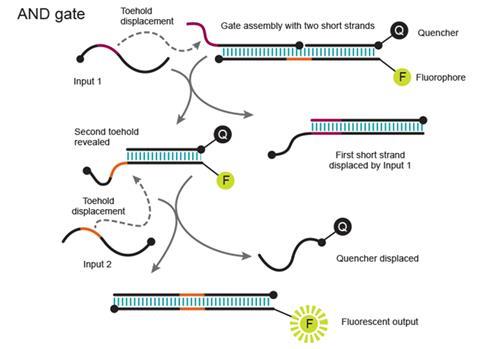
Winfree观察到,这有助于最大限度地减少不必要的“串扰”,串扰是由困扰Adleman方法的不正确的DNA配对引起的。他强调,化学系统不像电子系统,在电子系统中,信号是由互不干扰的独立电线上的电子传递的。相反,信号以分子的形式漂浮在溶液中,随机碰撞在一起。将信号作为序列送入与门,只能进行一个特定的位移,这接近于通过导线实现的分离。
然而,在链置换中,使DNA数字化并不止于此。Winfree解释说:“如果你有一个错误的信号,它应该是一个高浓度的数字‘信号’,但它只是一个中高浓度,你需要放大它。”“当问题是不需要的信号时,隔离所需信号的一种方法是让少量竞争者消耗不需要的信号。所需的信号就可以通过电路传播。在正确的信号通过竞争对手后,你需要一个催化反应,将剩余的信号放大到标准值。
2011年,Winfree和同事露露钱已经产生了一个电路,荧光可以报告15的平方根,四舍五入到最接近的整数。5该电路将几个链位移逻辑门组合在一起,每个逻辑门由几个寡核苷酸组成,整个电路包含130个不同的链。Winfree和Qian从四种不同荧光输出产生的颜色中读出二进制结果。Winfree强调:“在每一个AND或or门上,都有一个竞争过程,然后是催化放大。”
Soup-er电脑
在英国剑桥的微软研究院,安德鲁•菲利普斯他的同事建立了一种编程语言叫做DSD用于设计像Winfree团队那样的“DNA链置换”系统。它与一种称为化学反应网络(CRNs),模拟分子成分如何相互作用。Phillips将其比作在PC上编程,只是将指令体现在DNA中,而不是CD或硬盘驱动器上。

“在个人电脑上,你用C之类的语言写程序,然后把它编译成某种中间语言,然后把它编译成计算机可以执行的0和1。DNA就像0和1,crn是中间语言,在这之上是DSD。这使我们能够提出链位移算法,编译crn,然后用分子构建这些crn。设计算法是一个挑战。典型的计算机程序就像一个连续的食谱,而化学程序更像一碗汤。”
2013年,微软研究团队与两位美国学者一起使用DSD:大卫Soloveichik加州大学旧金山分校,以及Georg Seelig在西雅图的华盛顿大学。这两个团队一起从零开始设计和建造了可以与DNA传感器和电机一起工作的链位移控制模块电路。6为了展示利用链位移(Soloveichik和Seelig几年前作为Winfree加州理工学院团队的一员帮助发明了链位移)可以完成什么计算,一个控制器实现了“近似多数”算法。
该算法将两个可能的输出链的种群反应在一起,这样它们就可以通过形成可以放大输出的中间产物来“商定哪个种群更大”。这种结构消耗了两条信号链,有效地相互抵消,最终导致浓度较高的一条完全占主导地位。菲利普斯指出,同样的算法用于跨网络系统的计算,以及细胞分裂时的计算,尽管在DNA编程中仍然很慢。他承认:“这个算法需要15个小时。”你不会有一部运行这些算法的手机。它们的优势在于可以在细胞内运行。”
让DNA变得更好?
Soloveichik和Seelig不是通过化学合成寡核苷酸,而是通过将DNA插入其中来制造电路大肠杆菌的基因组,培育细菌,然后再次提取它们的DNA。Phillips说,这以相当的成本克服了由错误合成序列引起的另一个信息损失来源。
Winfree称这是“非常令人印象深刻和重要的”,特别是对于“模拟”计算,它允许数值在数字计算指定的1和0之间。但是,虽然细菌产生了功能更好的电路,但化学合成的寡核苷酸中的错误“还不是交易的破坏者”。“在我的实验室里,我们设计了一个序列,给一家公司发邮件,几天后他们就会把DNA发给我们。我们的典型实验可能使用价值100美元(66英镑)的试剂。我们不关心他们是怎么制造的,我们关心的是它不会扰乱实验。但我不认为这种综合是一个主要障碍。
事实上,在利用DNA制造更复杂的分子计算机的道路上,Winfree没有看到任何不可逾越的障碍。他的合作者已经设计并制造了一个由DNA、RNA和酶组成的生化振荡器“时钟”,并将其放置在油滴中,充当简单的“人造细胞”。7Winfree解释说,这是向“分子机器人”迈出的一步。“我们想要一种像细胞一样小的设备,它可以在化学环境中导航,释放出与环境相互作用的化学物质,并具有化学传感器来查看其中的情况。”他说,利用DNA计算来提供必要的控制,让系统在正确的时间做正确的事情,这就是分子编程。

编程DNA系统的进展,可以直接与生物化学物质相互作用,甚至改变了艾灵顿。他说:“我们正在做一件非常简单的事情,在分子诊断中过滤噪音。”8“一个问题是,如果你放大了一个目标链,你也放大了不需要的链。如何从噪声中过滤信号?“一个答案是利用链位移进行基本DNA计算的电路。在滤除这些噪声时,该方法的选择性与防止Winfree电路中不同信号之间的串扰功能类似。
艾灵顿现在和Paratus诊断该公司将这项技术商业化应用于可以从唾液或尿液中的RNA或小分子中诊断疾病的设备。艾灵顿希望它能使“家庭医生”产品成为现实,但对成功的可能性非常现实。他笑着说,我不担心市场会在不久的将来告诉我它的真实想法。
然而,他补充说,DNA编程并不适合某些可能的应用。“我与笨重的DNA药物输送装置划清界限——它们完全站不住脚。它们的成本太高了——该领域面临的是成本低廉但疗效显著的小分子。”
改变生活的结果
彭殷来自美国波士顿哈佛医学院威斯研究所的他也认为诊断方法很重要,但他有一个更远大的长期愿景。他说:“你可以设想一种纳米设备,在检测到蛋白质或RNA分子等疾病特征时,启动蛋白质的生产,通过改变患病细胞的颜色来诊断疾病,或者通过杀死细胞来治愈疾病。”2014年10月,Yin的团队更接近这一场景,发表了从头设计的RNA电路,控制蛋白质合成大肠杆菌细胞。9

科学家们利用链位移来提供低串扰水平,以使电路在细胞中发挥作用,并创建一个强大的设计框架。当同行评审人员要求进行大量额外的实验时,这一点有所帮助。殷回忆道:“我们当时说,‘我们的系统已经优化过了,所以我们也许能做到这一点。’”领导这项工作的博士后亚历克斯·格林(Alex Green)花了两年半的时间制定了基本原则。他在三个月内完成了额外的实验。格林和他的同事们还能够在“短短几天”内将这些知识转化为纸上的埃博拉传感器。10殷兴奋地说:“这显示了卓越的可编程性和鲁棒性,这对合成生物学来说是令人兴奋的。”他透露,虽然纸张传感器仍处于早期阶段,但许多生物技术公司都对它们感兴趣。
尹的团队设计了大肠杆菌自己产生两种RNA成分一个是“立足点开关”RNA环,包含蛋白质制造的指令,在第二个“触发”组件存在时打开。一旦环被打开,核糖体就可以结合,读取它,并制造它编码的蛋白质。虽然这是简单的如果/那么逻辑-如果检测RNA A然后制造蛋白质B - Yin强调了它的力量。以前的RNA系统从自然界中进化而来,已经有了类似的功能,但通常在开启和关闭状态下,蛋白质浓度之间的差异相对较小,只有50倍。平均而言,在新体系中,这一差距是400倍。
尹强调,这证明了理性设计的力量。“令人惊讶的是,这些合成的基因调节器比那些进化优化的调节器要好得多,”他兴奋地说。“进化起作用,但物理学起主导作用。这是我们在试管中开发的分子编程原理可以应用于活细胞的第一次实质性演示。但这只是一个起点。我们正在进行的工作将证明比以往任何时候都更复杂的体内计算。”
在计算方面,DNA和RNA的能力仍然很原始。但分子编程在与生物系统交互方面的能力已经很明显,可以用于疾病测试、制造分子机器人,甚至修饰活细胞。受到破解人与人之间安全电子通信的潜力的启发,DNA计算和分子编程现在正在帮助破解也许是所有生命中最大的谜团。
安徒生(@andyextance)是一位生活在英国埃克塞特的科学作家










暂无评论