IBM模型还可以找到在不同网站的目标蛋白质分子结合
一个人工智能(AI)模型已经确定了潜在的抗病毒分子抑制Sars-CoV-2病毒,从目标蛋白质的序列信息,没有任何知识的蛋白质的三维结构或已知的结合位点。
英国牛津大学的研究人员来自IBM和,训练一个叫做CogMol生成AI, IBM的一部分MolFormer-XL使用通用信息模型中,蛋白质和他们的绑定属性。然后他们使用这一模型来识别潜在的抗病毒药物Covid-19病毒。的建议是使用IBM的缩小RXN神经网络识别那些可能合成。最有前途的然后由合同制造商烯胺在乌克兰,对病毒和测试。(花)只有几周的最少的知识想出新的抑制剂新型病原体,”解释道Payel DasIBM的人工智能科学部门的经理。
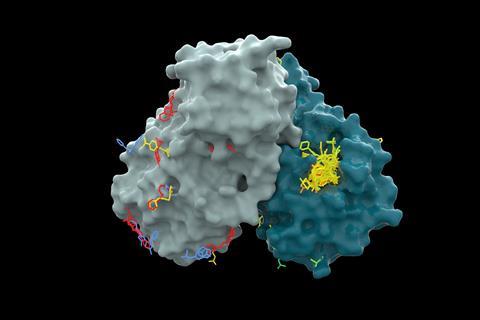
结合CogMol与其他人工智能算法允许团队专注数以千计的初步建议对分子药物类属性,以及他们的能力很快合成。化学家在烯胺然后选择8—4主要针对每个峰值和蛋白酶。在实验室测试中,两个从每组四个成功抑制各自的目标蛋白质。Das表明,即便是这种温和50%的成功率,给定输入速度和简单的模型意味着这类技术可以帮助人类探索化学世界”和快速评估各种可能性。的你也会发现这种研究可以识别病原体针对这部小说的新机制。
IBM研究人员提示CogMol Sars-CoV-2飙升的蛋白质序列和主要蛋白酶蛋白试图对可能的抗病毒药物。而其他人工智能程序需要结构性的数据或陈述的分子预测它们的属性,CogMol输入只是简单的文本字符串相关的蛋白质的序列。的方法没有结构预测,也不画推断绑定口袋或其他物理图案,”说詹森•克雷恩一个IBM高级研究科学家和牛津大学客座教授。
以及选择的分子合成,研究小组分析了CogMol许多其他建议的计算,来确定他们在哪里以及如何可能码头到目标蛋白质的结构模型。他们发现分子可以绑定到相对优雅的网站上的蛋白质。我们很惊讶知道分子不攻击目标点(通常),(受体结合域)的蛋白质,”达斯补充道。“我们甚至不知道这都是不可能的。
Das和科伦解释,替代结合位点抗病毒药物——远离蛋白质绑定其生物目标——可能不太容易突变病毒进化,这可以减少新的抗病毒药物需要跟上突变。研究人员希望能继续进行试验,这些分子实验室探索他们的抗病毒能力。
像牛津建立了大流行科学研究所2021年Das,克雷恩和其他IBM研究人员希望MoLFormer-XL CogMol及其伞软件应用于其他病毒作为更广泛的计划的一部分。(研究所)是加紧创建这个强烈自律连接环境中人工智能技术,和物理建模,都集中在一起,”科伦说。“这[研究]是一个议程setter(未来的合作)。
IBM已经在4月与制药公司合作建立了现代化2023年。现代化是寻找新的抗病毒药物对严重急性和非典型肺炎和其他病毒,所以使用IBM的CogMol软件可能会加速这一过程。与现代化,我们使用整个MolFormer套件,包括CogMol帮助他们设计新的核糖核酸疫苗稳定器,”达斯补充道。
引用
V Chenthamarakshan等,科学。睡觉。,2023,DOI:10.1126 / sciadv.adg7865













还没有评论