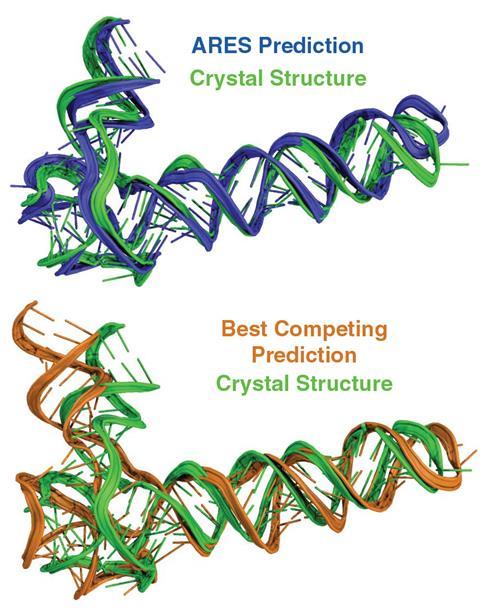
一个由生物化学家和计算机科学家组成的团队开发了一种新方法,通过使用少量已知RNA形状训练的人工智能系统,准确预测RNA分子的三维结构。
专家们称赞这一进展是对计算预测RNA结构挑战的重大改进,并表示这可能会导致更好地理解RNA在细胞功能和新的治疗药物中的作用。
Rhiju Das加州斯坦福大学生物化学副教授,他说新的机器学习系统——被称为原子旋转等变记分器(Ares)——使用“等变”神经网络来准确区分RNA分子的三维结构。
Das解释说,等变神经网络中的计算“神经元”不仅像其他类型的神经网络一样使用数字来激活,而且还使用向量、张量和其他类型的可量化对象。这使得Ares能够评估RNA分子的结构基元,例如不同类型的螺旋、“发夹”和茎,这种方法被称为“几何深度学习”。
基本训练
研究人员仅用18种精心设计的rna训练Ares系统,这些rna的结构是经过艰苦的实验确定的。该系统随后在列表中列出的更大的RNA结构上进行了测试RNA-Puzzles网站这是一项已有十年历史的科学竞赛。
他们使用罗塞塔分子建模软件的一个版本,从网站上为六种解决的RNA生成了1500多个不同的结构模型,同时确保至少1%的模型是“接近原生的”——这意味着它们与RNA的真实结构密切对应。
然后,他们使用Ares计算每个模型结构的得分,并使用Rosetta软件的评分功能、核糖核酸统计协议(Rasp)和3dRNAscore计算得分。Ares系统在其他三个评分功能上的表现明显优于其他三个评分功能:在其10个得分最高的模型中,有81%包含了至少一种“接近原生”的模型结构,而Rosetta为48%,Rasp为48%,3dRNAscore为33%。
Ares在测试中也超越了其他评分功能,测试池中没有“接近原生”的模型。它还在四轮rna谜题比赛(rna的真实结构尚不清楚)中表现出色,在每种情况下提交的模型中都是最准确的。
达斯说:“令人惊讶的是,我们能够从如此少的训练样本中训练战神网络,然后在rna谜题盲竞赛中获得最先进的结果。”
迎头赶上
研究人员写道,关于RNA结构的科学知识远远落后于蛋白质结构的科学知识,而蛋白质结构得益于人工智能预测系统AlphaFold来自谷歌的子公司DeepMind。相比之下,它们通常是在包含数千个结构的庞大数据集上进行训练的。
研究人员写道:“转录为RNA的人类基因组的比例大约是编码蛋白质的30倍,但可用的RNA结构的数量不到蛋白质的1%。”这主要是因为相关RNA的结构比蛋白质的结构更不容易为人所知,因此不能用作模板。
他们现在希望Ares开创的几何深度学习方法将有助于刺激对RNA结构的研究,尽管到目前为止,它只解决了过程的一部分。达斯说:“我们的论文仍然依赖于用上一代罗塞塔软件生成的模型池,这些软件没有使用神经网络。”“现在使用几何深度学习的技巧来生成RNA 3D模型将是非常棒的。”
由于Ares只需要原子坐标和化学元素作为输入,同样的方法可以应用于涉及三维化学结构的其他领域。类似的等变神经网络在最近的研究论文中已经成功地使用AlphaFold和Rosetta软件达斯说。
计算生物学家亚历克斯·贝特曼没有参与这项研究的欧洲生物信息学研究所(European Bioinformatics Institute)的教授指出,RNA结构的预测已经落后于AlphaFold在蛋白质结构预测方面取得的进展。但他说,“战神的发展已经在该领域迈出了一大步,我们期待着获得这些模型的使用权。”
他警告说,“战神”的精度仍有待提高。他说:“也许,受到AlphaFold 2.0方法发布的启发,我们将在未来几个月和几年内看到更好的方法和模型。”“这是RNA研究的一个非常激动人心的时刻。”
参考文献
RJL汤森等,科学, 2021,373, 1047 (doi:10.1126 / science.abe5650)





暂无评论